海洋细菌酶混杂催化功能的定向进化研究取得进展
中国科学院热带海洋生物资源与生态重点实验室藻类资源与生态工程研究团队在工程化改造酶蛋白的混杂催化功能方面取得新进展,相关研究论文“Repurposing a bacterial prolidase for organophosphorus hydrolysis: Reshaped catalytic cavity switches substrate selectivity”于2020年6月27日发表于《Biotechnology and Bioengineering》。杨键副研究员与肖运柱博士是论文共同第一作者,龙丽娟研究员是通讯作者。
酶催化生物体内化学反应,是维持生命代谢有序运转的重要驱动力。传统观点认为酶催化化学反应是非常精确专一的,然而近年越来越多的研究表明酶具有多种“兼职”功能,这种酶催化功能的非特异现象称为混杂性(Promiscuity)。酶的催化混杂性可为生物提供“兵器库”,帮助生物适应多变的化学环境。在工业界,开发利用酶的混杂催化功能可帮助人们合成化学分子、修复污染环境。
该研究发现一种海洋细菌脯氨酸二肽酶具有混杂水解对氧磷农药的功能,该酶水解二肽的活性(kcat/Km=(7.68±1.24)×104 M-1s-1)是水解对氧磷(kcat/Km=(0.94±0.09)×103 M-1s-1)的近100倍。作者期望通过优化底物结合口袋提高该肽酶水解对氧磷的活性,对组成该酶底物结合口袋的11个氨基酸残基进行了定点饱和突变和组合突变。经过多轮筛选从4000余个突变体中获得最优突变D45W/H226G。相较野生型,D45W/H226G水解对氧磷的活性提高了30倍(kcat/Km=(2.96±0.05)×104 M-1s-1),且其对其他有机磷类化合物的底物谱也获得显著拓宽。有趣的是,伴随着混杂活性的提高,天然肽酶活性显著下降(kcat/Km=1.76±0.02 M-1s-1),突变酶与野生酶的催化选择性发生106倍转换(图1)。为探究两个氨基酸残基突变造成该酶底物选择性差异的原因,作者利用X-射线衍射技术解析了野生酶与突变酶的蛋白晶体结构。通过比较蛋白结构,结合分子动力学模拟计算揭示了底物结合口袋形态的变化影响两种底物与酶活性中心的结合模式,使对氧磷在突变酶中的结合构象更利于催化反应进行,而二肽底物在突变酶中的结合构象则刚好相反(图2)。
该研究改良利用酶的混杂催化功能,将细菌肽酶体外定向进化为对氧磷水解酶,为有机磷污染物的微生物降解提供了高效酶元件,也有助于提升酶催化底物选择性分子层面的认识。
中国科学院战略性先导科技专项(XDA13020301)、南方海洋科学与工程广东省实验室(广州)人才团队引进重大专项 (GML2019ZD0404)和广州市科技计划项目(201904010165)共同资助了该研究。
相关论文链接:https://doi.org/10.1002/bit.27455
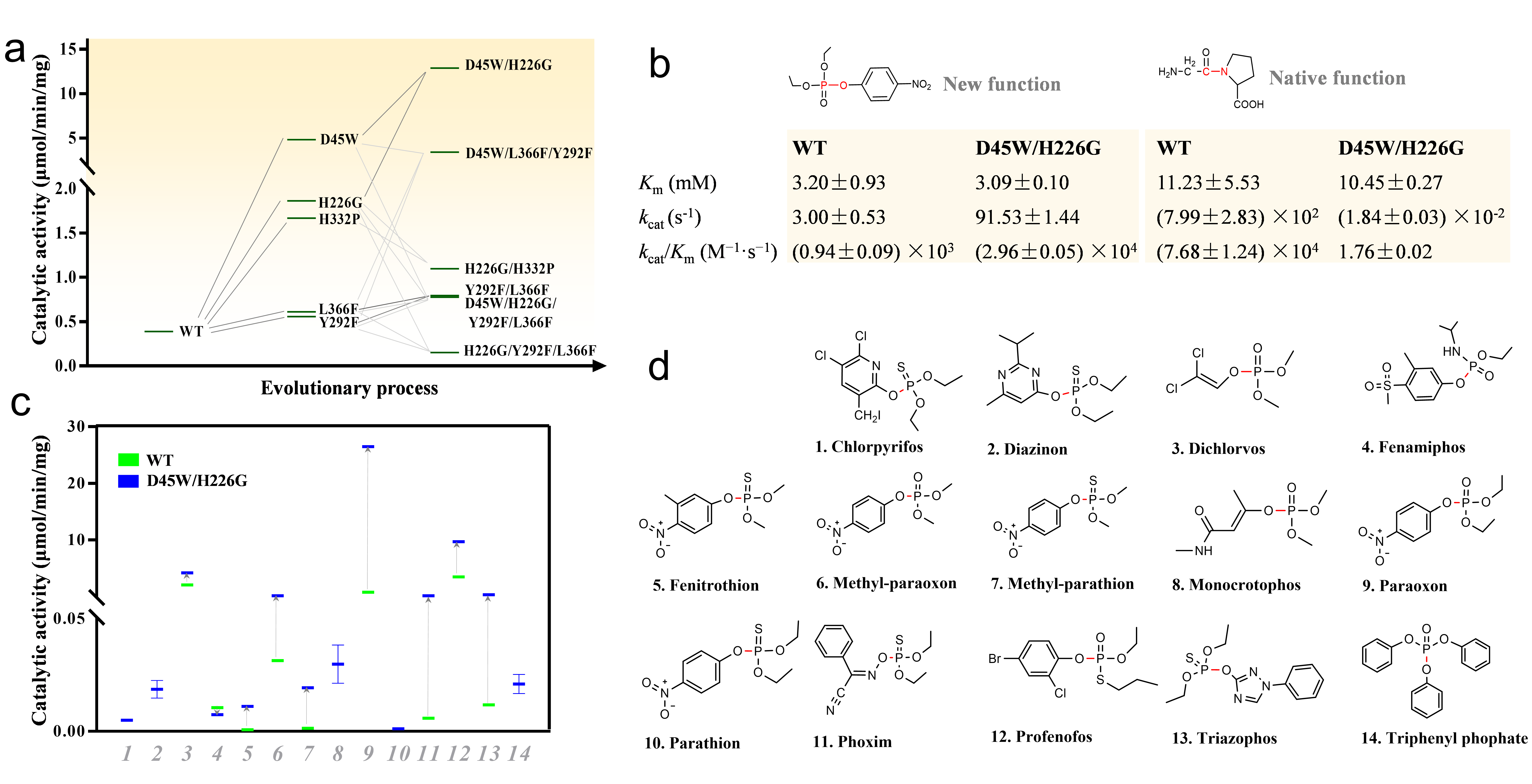
图1 (a)海洋细菌肽酶的体外定向进化过程;(b)野生酶与突变酶的催化动力学参数;(c)野生酶与突变酶对不同有机磷水解活性的比较;(d)有机磷类底物的化学结构
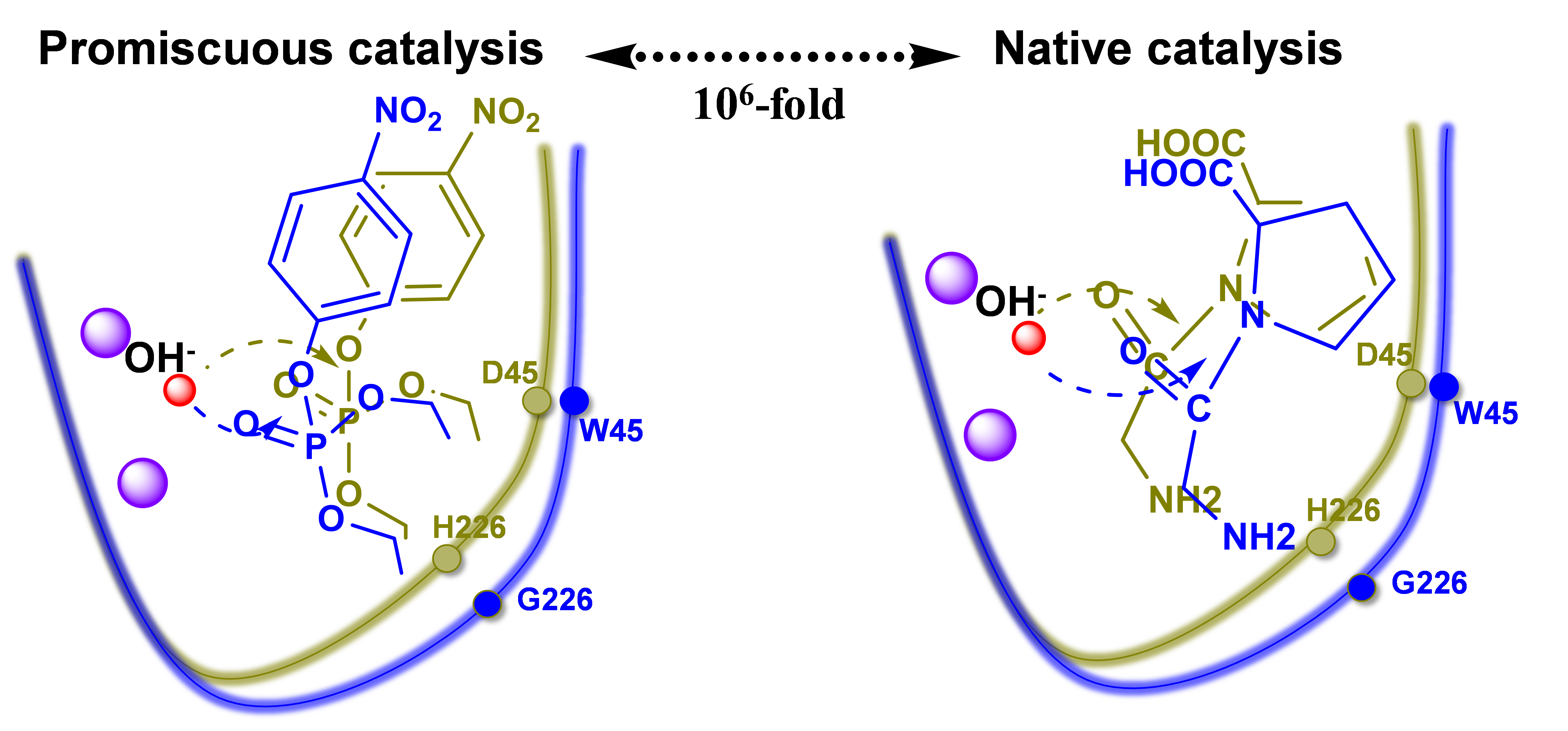
图2 底物选择性转换机制示意图
附件下载:
 粤公网安备44011502001245号
粤公网安备44011502001245号