科研人员建立原噬菌体de novo预测新算法
中国科学院南海海洋研究所研究员王晓雪团队建立了一种不依赖于噬菌体基因序列相似性的原噬菌体de novo预测新算法,集成分析流程的工具名为Prophage Tracer。相关研究9月22日在线发表于《核酸研究》。汤开浩博士为论文第一作者,王晓雪为通讯作者。来自天津生物芯片技术有限责任公司的孙亚民博士也参与基因组分析工作。
烈性噬菌体侵染细菌宿主后,大量繁殖,裂解宿主细胞释放子代噬菌体粒子。温和噬菌体则是进入宿主细胞内,将自身的DNA整合在宿主的基因组中,随着宿主细胞的复制而复制。这个过程称为噬菌体的“溶原化”,整合在宿主基因组中的噬菌体DNA称为“原噬菌体” (prophage)。特定条件能重新激活原噬菌体使其裂解宿主。目前研究发现在人、小鼠肠道以及珊瑚微生物组中溶原化的温和噬菌体比裂解状态更加普遍。温和噬菌体的溶原-裂解转换是微生物生态领域的重要科学问题之一。
温和噬菌体能和宿主形成长期共生关系,其整合切离等可以破坏或恢复宿主的基因正常功能,是宿主基因表达调控的一种重要途径。研究团队的前期研究发现希瓦氏菌的原噬菌体CP4So可以在低温诱导条件下发生切离,是细菌一种重要的适冷机制(1),并且 CP4So的切离受到宿主温度依赖性的H-NS磷酸化调控(2)。因此精确鉴定细菌中的原噬菌体及其插入位点对于研究温和噬菌体与宿主的共生关系至关重要。目前鉴定原噬菌体的方法主要依赖利用已知的噬菌体进行序列相似性检索。但是由于噬菌体的基因组变异快,基于序列相似性检索方法很难发现未知类型噬菌体。此外,对噬菌体插入位点的预测也不准确,很难区分原噬菌体携带的”cargo基因”和宿主基因的边界。
为了解决上述问题,汤开浩等建立了一种从头预测原噬菌体的方法(图1)。该方法主要利用原噬菌体整合切离过程中会产生基因组结构变异基因组序列。这些序列隐藏在细菌基因组、转录组测序数据的reads中。通过建立重叠的序列比对方法,追踪和挖掘埋藏于测序原始数据中reads。只需甄别到1-2条reads就能精确定位原噬菌体(精确到单个碱基)。由于该方法不依赖于序列相似性,因此能够预测到未知的噬菌体。通过挖掘,成功的在珊瑚共附生细菌中鉴定到九个温和噬菌体,其中两个为新颖的温和噬菌体。
本研究工作得到国家杰出青年科学基金、基金委水圈微生物重大研究计划重点项目、面上项目、国家重点研发计划等项目的共同资助。
相关论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab824/6374144
Prophage Tracer下载地址:https://github.com/WangLab-SCSIO/Prophage_Tracer
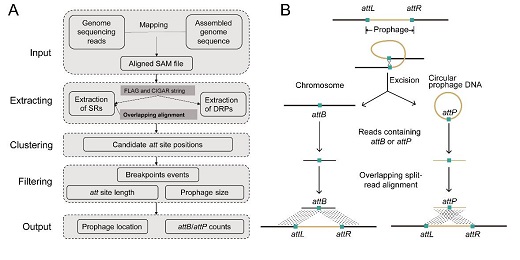
图1 Prophage Tracer流程
附件下载:
 粤公网安备44011502001245号
粤公网安备44011502001245号