链霉菌天然产物沉默基因簇激活方法开发取得新进展
近日,中国科学院南海海洋研究所热带海洋环境与岛礁生态全国重点实验室、广东省海洋药物重点实验室科研人员在链霉菌天然产物沉默基因簇的激活方法开发中取得新进展,相关成果以“Development of A Salt-Enhanced Promoter Strategy for Activating Silent Biosynthetic Gene Clusters from Streptomycetes”为题发表在代谢工程领域著名期刊Metabolic Engineering(代谢工程)。中国科学院南海海洋研究所博士王利娟、博士朱梦奕和副研究员杨春芳为本文共同第一作者,研究员张长生为本文通讯作者。
链霉菌天然产物是药物研发的重要资源。近年来,随着测序成本的降低和生物信息学工具的普及,大量未知天然产物的生物合成基因簇序列被解析公布,亟待深入挖掘。然而,当前基因组挖掘面临两大核心挑战:一是链霉菌生物合成基因簇在实验室条件下大多数呈沉默或低表达状态,其产物丰度极低;二是部分野生型链霉菌无法进行遗传操作,导致其天然产物生物合成途径挖掘受限。
针对链霉菌基因组挖掘中的上述瓶颈问题,研究团队近期开发了一种基于盐响应启动子的新型挖掘策略。该策略可有效激活链霉菌天然产物沉默基因簇,并成功应用于三种活性天然产物的激活与高产。研究团队在对南海来源的、含有丰富天然产物生物合成基因簇但无法进行遗传操作的链霉菌开展基因组挖掘的过程中,采用“异源表达-细菌人工染色体-启动子工程”的协同策略对两个沉默的大型天然产物生物合成基因簇进行激活。意外发现在培养基添加钠盐或钾盐,尤其是添加1% KCl,能够显著增强广泛使用的组成型启动子kasOp*的强度,进而高效激活其下游基因簇的表达并大幅提高目标天然产物的产量(图1)。
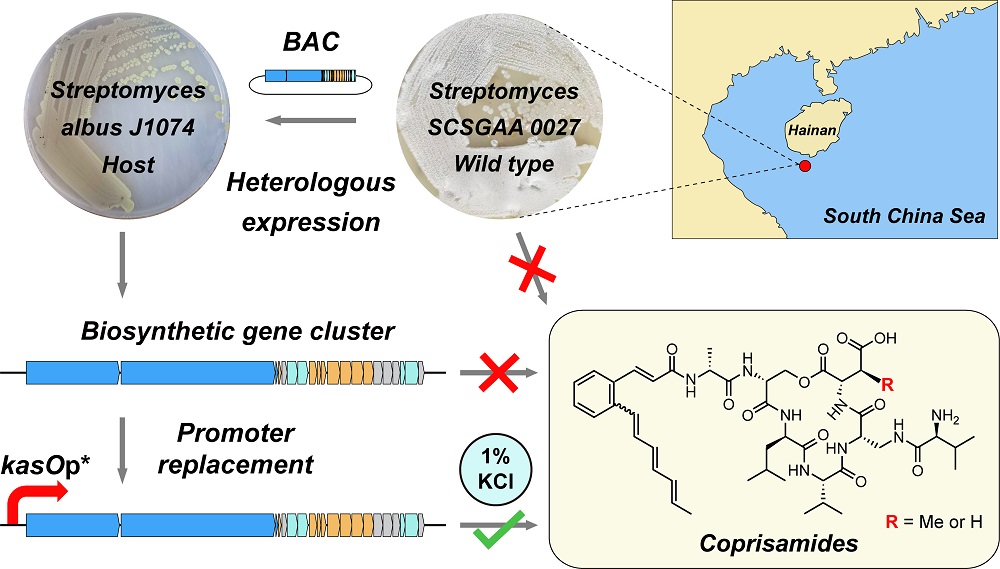 图1 采用kasOp*-KCl协同策略激活海洋链霉菌中沉默天然产物生物合成基因簇
图1 采用kasOp*-KCl协同策略激活海洋链霉菌中沉默天然产物生物合成基因簇
研究人员首次发现了行业通用的组成型启动子kasOp*的盐响应特性,同时通过实验证实kasOp*-KCl策略对不同培养基、不同基因簇及常见链霉菌异源表达宿主均具有普适性。采用这种策略使coprisamide(聚酮-聚肽杂合环肽)、padanamide(非核糖体多肽) 和 SF2768 (异腈酯肽)这三种活性天然产物的产量均达到目前报道的最高水平(图2)。这一发现凸显了这种基于遗传元件盐响应特性的便捷基因组挖掘方法,在激活链霉菌沉默产物生物合成及增强其产量方面的潜在实用价值,为链霉菌沉默基因簇的挖掘及天然产物增产研究提供了全新工具。
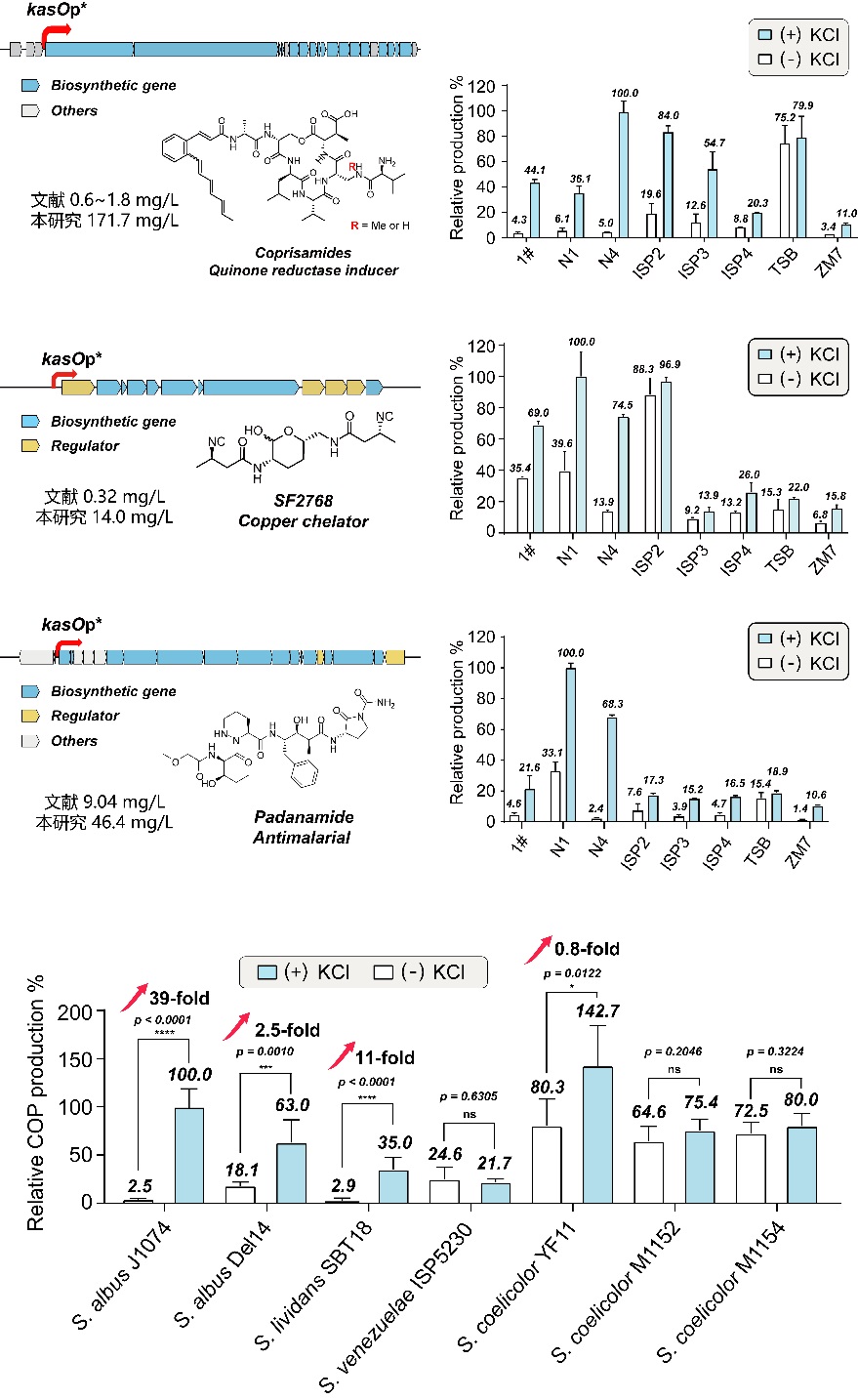 图2 KasOp*-KCl策略对不同天然产物、不同培养基和不同宿主的普适性评估
图2 KasOp*-KCl策略对不同天然产物、不同培养基和不同宿主的普适性评估
以上研究工作得到了国家重点研发计划“合成生物学”重点专项、国家自然科学基金面上项目、中国科学院青年创新促进会项目、博士后创新人才支持计划、广东省基础与应用基础研究基金项目、南沙区重点科技计划项目和海南省自然科学基金等项目资助。
论文信息:Lijuan Wang#,Mengyi Zhu#,Chunfang Yang#,Siqi Zhu,Bin Tan,Shu-Hua Qi,Yiguang Zhu,and Changsheng Zhang*. Development of A Salt-Enhanced Promoter Strategy for Activating Silent Biosynthetic Gene Clusters from Streptomycetes. Metabolic Engineering,2025,92:51-62.
论文链接:https://doi.org/10.1016/j.ymben.2025.07.007
附件下载:
 粤公网安备44011502001245号
粤公网安备44011502001245号