基于遗传图谱和QTL定位发现Polycystin基因参与牡蛎壳形成
中国科学院南海海洋研究所热带海洋生物资源与生态实验室(LMB)喻子牛研究员团队在牡蛎高密度遗传连锁图谱构建和壳生长QTL定位、功能基因验证方面取得重要进展,相关研究成果High-resolution genetic maps and QTL mapping applications reveal Polycystin gene involvement in oyster shell formation正式发表于国际综合期刊《iScience》。南海海洋研究所助理研究员马海涛、副研究员秦艳平等为论文共同第一作者,研究员喻子牛和研究员张跃环为论文通讯作者。
香港牡蛎和熊本牡蛎是华南沿海重要的经济优势牡蛎种,团队成功进行了两种牡蛎间的人工远缘杂交。本研究以两种牡蛎正反交家系为材料,构建了它们的高密度遗传连锁图谱。正交家系中母本(香港牡蛎)获得了10个连锁群,与其染色体数一致,标记间的平均间隔为1.5cM,图谱总长度为899.37cM,图谱的覆盖率为97.30%;父本(熊本牡蛎)也获得了10个连锁群,与其染色体数一致,标记间的平均间隔、图谱总长度、图谱覆盖率分别为1.22cM、836.94cM、97.40%。反交家系中母本(熊本牡蛎)获得了10个连锁群,与其染色体数一致,标记间的平均间隔为0.94cM,图谱总长度为1096.09cM,图谱的覆盖率为98.63%;父本(香港牡蛎)获得了10个连锁群,与其染色体数一致,标记间的平均间隔、图谱总长度、图谱覆盖率分别为1.23cM、653.36cM、97.04%。另外熊本牡蛎整合遗传图谱标记间的平均间隔达到0.75cM。
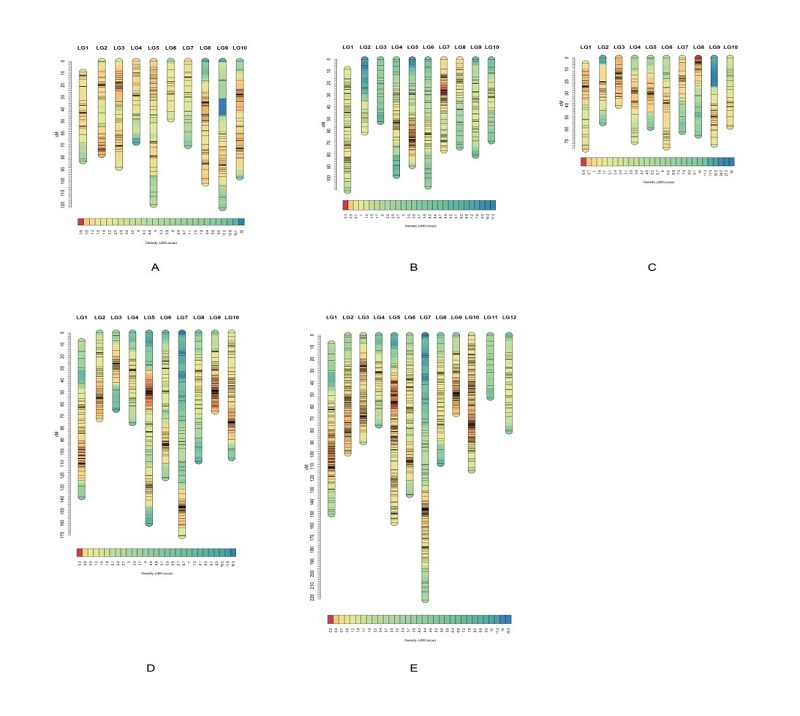
图1 高密度遗传连锁图谱
根据两个作图家系的表型数据和遗传连锁图谱,共定位了35个与壳生长相关QTL位点,可解释的表型遗传变异为6.1-26.4%(壳高)、4.9-20.0%(壳长)。通过基因组基因比对,在QTL上共鉴定出22个候选基因,其中Polycystin基因具有最大LOD值和最高可解释的表型变异(26.4%-壳高/20.0%-壳长)。
该成果在两种牡蛎中首先克隆了Polycystin基因的全序列;q-PCR发现在牡蛎外套膜中表达水平最高,破壳试验中其表达水平随着贝壳形成过程发生变化,原位杂交也发现该基因表达于外套膜外褶和中褶边缘区域;不同发育阶段的定量表达分析,发现在两种牡蛎中从受精卵开始表达量逐渐升高并在囊胚期达到最高峰,该基因可能参与了牡蛎壳早期发育的钙离子储存,为担轮幼虫及后期牡蛎壳发育提供基础;RNAi技术成功抑制该基因表达后,通过扫描电镜发现新生贝壳出现矿物层沉积不规则的现象。综上所述,Polycystin基因很可能通过介导钙离子运输的方式参与了牡蛎壳形成。
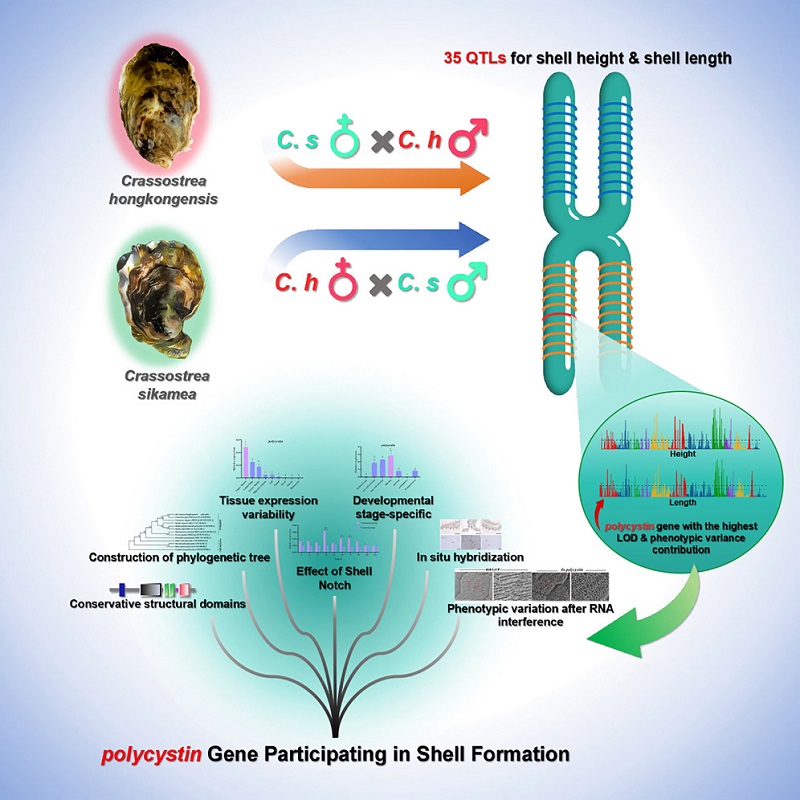
图2 功能验证Polycystin基因参与了牡蛎壳形成
本研究首次构建了香港牡蛎和熊本牡蛎的高密度遗传连锁图谱并定位了壳生长相关QTL位点,并证明Polycystin基因参与了牡蛎壳形成,研究结果将为两种牡蛎的分子育种提供理论基础和技术支撑。
该成果得到了广东省自然科学基金、国家自然科学基金、国家贝类产业技术体系、科技部重点研发计划、广东省重点研发计划、海南省重点研发计划和广州市重点研发计划等项目的联合资助。
相关论文信息:High-resolution genetic maps and QTL mapping applications reveal Polycystin gene involvement in oyster shell formation. iScience,2025,28: 113986.
原文链接:https://doi.org/10.1016/j.isci.2025.113986
附件下载:
 粤公网安备44011502001245号
粤公网安备44011502001245号